R言語でフィッシャーの正確率検定を実行する方法をみていきます。
前回紹介したカイ2乗検定の引き続き、カテゴリカルデータの検定方法であるフィッシャーの正確率検定の実行例を紹介します。
カイ2乗検定については以下の記事を参照してください。
-

【R言語】カイ二乗検定 適合度検定・独立性の検定
R言語でカイ2乗検定を実行する方法を解説していきます。データセットの用意から検定までの一連の流れを紹介していきます。 カイ2乗検定の種類として適合度検定や独立性の検定がありますが、R言語に標準で実装さ ...
続きを見る
この記事で扱うプログラミングコードは以下に保存してあります。
フィッシャーの正確率検定
仮説検定
フィッシャーの正確率検定は\(r\times c\)の分割表について、2つの分布(分割表の行に関する分布と列に関する分布)は統計的に独立であるかを検定します。
フィッシャーの正確率検定
| 男性 | 女性 | 行の総計 | |
| 効果あり | a | b | a+b |
| 効果なし | c | d | c+d |
| 列の総計 | a+c | b+d | 総計 n=a+b+c+d |
ここに\(a, b, c, d\)は分割表の各セル数を表します。今、男性と女性を表す分布と効果ありと効果なしの分布は統計的に独立であるかを検定したいとします。この仮説検定のp値は、次の超幾何分布の累積確率を用います。
この確率は\(n\)個から\(a+c\)の男性を選んだ時に、効果ありの群から\(a\)個、効果なしの群から\(c\)個抽出す確率となります。(aは超幾何分布に従う)。p値はセル数\(a\)がより極端な場合(この場合は\(a\)がより少ない場合)での上記の確率の和となります。
効果を表す周辺分布の確率をそれぞれ\(p_A, q_A\)とし、性別を表す周辺分布の確率をそれぞれ\(p_B, q_B\)とすると、\(a, b, c, d\)の各セルに対応する確率はそれぞれ\(p_Ap_A, p_Aq_B, q_Ap_B, q_Bq_B\)となります。\(a+b\)と\(c+d\)が与えられた下で、仮に\(a\)が\(np_Ap_A\)に近い場合、その確率とそれより極端な事象の確率\(p\)の和は大きくなり、逆に、\(a\)が\(np_Ap_A\)に近くない場合、その和は小さくなることが分かります。
関数fisher.test
フィッシャーの正確率検定を行うには関数fisher.testを用います。以下、関数fisher.testとその引数の説明となります。
fisher.test(x, y = NULL, workspace = 200000, hybrid = FALSE, hybridPars = c(expect = 5, percent = 80, Emin = 1), control = list(), or = 1, alternative = "two.sided", conf.int = TRUE, conf.level = 0.95, simulate.p.value = FALSE, B = 2000)
| x | 分割表を表すnumeric型の行列か、factor型のベクトル。 |
| y | xと同じ長さのfactor型のベクトル。xが行列である場合無視される。 |
| workspace | ネットワークアルゴリズムに用いられるワークスペースのサイズを決める整数。 |
| hybrid | TRUEの場合、正確率の代わりにハイブリッド近似を用いる。 |
| hybridPars | カイ2乗近似のためのコクラン条件を指定する長さ3のベクトル。 |
| control | low level algorithm controlのための要素をもつリスト。 |
| or | 帰無仮説で与えるオッズ比。 |
| alternative | "two.sided"で両側検定、"greater"で右片側検定、"less"で左片側検定を指定できる。 |
| conf.int | TRUEの場合、オッズ比の信頼区間が計算される。 |
| conf.level | 信頼区間に用いた信頼水準を結果に含める。 |
| simulate.p.value | TRUEの場合、モンテカルロシミュレーションを用いてp値を計算する。 |
| B | モンテカルロ法での試行回数。 |
この後、fisher.testの使用例を紹介します。データセットを用いて検定の一連の流れを見ていきます。
fisher.testの使用例
2×2分割表の場合
早速fisher.testを使用していきます。
まず検定に用いる分割表を作成する必要があります。
データセットとして上で紹介した効果と性別から成る分割表を作成します。関数matrixを用いることで2×2の分割表を作っています。
1 2 3 4 5 6 7 8 | > EffectGender <- matrix(c(1, 9, 7, 3), nrow = 2, + dimnames = list(Effect = c("効果あり", "効果なし"), + Gender = c("男性", "女性"))) > EffectGender Gender Effect 男性 女性 効果あり 1 7 効果なし 9 3 |
EffectGenderには5未満のセルが複数あることが分かります。セルの標本数が少ないため、カイ2乗検定ではなくフィッシャーの正確率検定を用いた方がよいことが言えます。
(r×cもありますが、簡便のため2×2の場合について考えています。)フィッシャーの正確率検定を実行するには、次のように引数xに分割表を代入します。
1 2 3 4 5 6 7 8 9 10 11 12 13 | > testResult <- fisher.test(EffectGender) > testResult Fisher's Exact Test for Count Data data: EffectGender p-value = 0.01977 alternative hypothesis: true odds ratio is not equal to 1 95 percent confidence interval: 0.0009621944 0.7209145117 sample estimates: odds ratio 0.05788421 |
上の結果から、p値=0.01977<0.05であり「効果は性別によって差がある」ことが言えました。
また、補足としてカイ2乗検定を実行すると、セルの標本数が少ないために「カイ二乗近似が正確ではありません」という警告が出ます。
1 2 3 4 5 6 7 8 9 | > chisq.test(EffectGender) #カイ二乗検定を行うと警告が出る Pearson's Chi-squared test with Yates' continuity correction data: EffectGender X-squared = 5.2083, df = 1, p-value = 0.02248 警告メッセージ: chisq.test(EffectGender) で: カイ自乗近似は不正確かもしれません |
次に検定結果について説明します。次のようにtestResultのうしろに$を付けることでオッズ比やp値を参照することができます。
1 2 3 | or <- unname(testResult$estimate) #オッズ比 ci <- unname(testResult$conf.int) #信頼区間 pValue <- unname(testResult$p.value) #p値 |
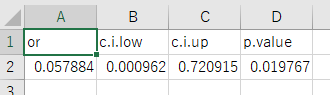
最後に、これらの結果をcsvファイルに出力していきます。オッズ比、オッズ比の信頼区間、p値を格納したデータフレームを作成し、resultTableに代入します。
1 2 3 4 | > resultTable <- data.frame(or = or, c.i.low = ci[1], c.i.up = ci[2], p.value = pValue) > resultTable or c.i.low c.i.up p.value 1 0.05788421 0.0009621944 0.7209145 0.01976661 |
write.csvを用いてcsvに出力すると、作業ディレクトリに次の検定結果が出力されます。
1 | write.csv(resultTable, "フィッシャーの正確率検定.csv", row.names = FALSE) |
r×c分割表の場合
最後にr×c分割表の使用例を紹介します。2×2分割表の場合検定結果にオッズ比等が含まれていましたが、オッズ比やその信頼区間が算出されないという違いがあります。
では、早速実行していきます。関数glを用いて、1~3と1~4のレベルを持つfactor型のデータdataAとdataBを作ります。data.frameを用いてデータフレームに格納したら、tableを用いることで簡単に集計表を作成することが可能です。
1 2 3 4 5 6 7 8 9 10 11 | > dataA <- gl(4 ,3, 12) > dataB <- gl(3, 2, 12) > data <- data.frame(A = dataA, B = dataB) > table_AB <- table(data) > table_AB B A 1 2 3 1 2 1 0 2 0 1 2 3 2 1 0 4 0 1 2 |
r×cの場合にも2×2のときと同様に、検定を行うことが可能です。
1 2 3 4 5 6 7 8 | > testResult <- fisher.test(table_AB) > testResult Fisher's Exact Test for Count Data data: table_AB p-value = 0.4016 alternative hypothesis: two.sided |
p値=0.4016より、「分布Aと分布Bは統計的に独立である」ことが言えました。
r×cの場合、検定結果にオッズ比やその信頼区間が含まれていないのが分かります。
実際に、testResultの後ろに$を付けると、p.value等しかないことが確認できます。
1 | pValue <- testResult$p.value #p値(オッズ比等は算出されない) |
まとめ
R言語でフィッシャーの正確率検定を実行する方法をみていきました。
カイ2乗検定と同様に、分割表を引数に代入するだけで簡単に検定を行うことが可能です。
ぜひ、この記事を解析に役立ててください。


