一般化線形モデルを行う方法を解説していきます。
R言語で回帰分析を行う方法については、以下の記事で紹介しています。
-

【R言語】関数lmによる線形回帰 単回帰分析・重回帰分析 回帰分析で予測する
R言語の回帰分析の実行方法を紹介していきます。回帰分析の実行方法だけでなく、回帰分析により作ったモデルでテストデータの予測も行っていきます。 実行方法では、回帰分析の結果の見方やその抽出方法を詳しく解 ...
続きを見る
一般化線形モデルを用いることで、母分布が正規分布でなくても線形モデルを構築することが可能となります。
例えば、目的変数が0と1のような二値である場合でも、回帰(予測)することが可能となります。
また、この記事で紹介しているプログラミングコードは以下のリンクからダウンロードすることができます。

関数glm
R言語で一般化線形モデルを実行するには、関数glmを用います。
基本的な使いかたは回帰分析で用いたlmと変わりません。
lmとの違いとして、引数に誤差構造やリンク関数を指定する点があります。
関数glmは以下の引数から成ります。
1 2 3 | glm(formula, family = gaussian, data, weights, subset, na.action, start = NULL, etastart, mustart, offset, control = list(…), model = TRUE, method = "glm.fit", x = FALSE, y = TRUE, singular.ok = TRUE, contrasts = NULL, …) |
引数は以下の通りです。
| formula | 回帰に用いるモデル(式) | offset | オフセット項の指定 |
| family | 誤差構造とモデルに用いられるリンク関数。 | control | 適合のプロセスを制御するパラメータのリスト |
| data | 回帰に用いるデータセット | model | 返り値にモデルを含むかどうか |
| weights | prior weights. 事前に重みを与えたい場合数値ベクトルで与える。 | method | モデルを適合する際に用いるアルゴリズム |
| subset | 回帰する際に用いる観測値の部分集合 | x | 返り値に計画行列Xを含むかどうか |
| na.action | dataがNAを含む場合にとる行動。 | y | 返り値に目的変数yを含むかどうか |
| start | 回帰係数の初期値 | singular.ok | 計画行列Xが非正則の場合、回帰を行うか |
| etastart | 線形予測しの初期値 | contrasts | オプションのリスト。詳細はcontrasts.arg。 |
| mustart | 線形予測値の平均値の初期値 |
上で紹介した引数のうち、familyについて詳しく見ていきます。
引数familyを変更することで、誤差構造とそれに対応するリンク関数を変更することが可能となります。
誤差構造とリンク関数を以下にまとめました。
| family | link |
| binomial | "logit", "probit", "cauchit", "log", "cloglog" |
| gaussian | "identity", "log", "inverse" |
| Gamma | "inverse", "identity", "log" |
| inverse.gaussian | "1/mu^2", "inverse", "identity", "log" |
| poisson | "log", "identity", "sqrt" |
| quasi | "logit", "probit", "clolog", "identity", "inverse", "log","1/mu^2", "sqrt" |
| quasibinomial | "logit", "probit", "cauchit", "log", "cloglog" |
| quasipoisson | "log", "identity", "sqrt" |
例として、目的変数がc(1, 3, 6, 3, 9, 2, 3, 4)のような離散値である場合、母集団にポアソン分布</spanを指定します。
また、c(1, 0, 1, 1, 0, 1, 1)のような二値である場合、二項分布を指定します。
この後、より具体的な実行方法をみていきます。
glmの実行例
誤差構造がポアソン分布
まず、glmの使用例として、ポアソン回帰の実行方法についてみていきます。
誤差構造がポアソン分布に従う場合の回帰分析の実行方法を解説していきます。
簡単に言うと、目的変数が「1, 2, 3, 4」といった離散値であるときの回帰分析の手順について説明します。
ここでは、以下の線形モデルから成るポアソン回帰の実行方法について触れます。
リンク関数にlogを使っていることに注意してください。
ポアソン回帰
ポアソン回帰は次の線形モデルで表されます。
ここに\(\lambda_i\)は事象の発生回数の期待値であり、\(\boldsymbol{x}_i = (x_{1i}, \ldots, x_{ki})^T\)である。
では早速、ポアソン回帰の実行方法をみていきましょう。
データセットの用意
目的変数がポアソン分布に従う場合のglmの実行方法について解説します。
次のようなデータセットポアソン分布に従う列countsを持つデータを用います。
1 2 3 4 | counts <- c(18, 17, 15, 20, 10, 20, 25, 13, 12, 19, 16, 14) outcome <- gl(3, 1, 12) treatment <- gl(3, 3, 12) data <- data.frame(counts, outcome, treatment) |
次のように、dataは離散変数のcountsと「1, 2, 3」を水準に持つ因子型の列outcomeとtreatmentから成ります。
1 2 3 4 5 6 7 8 | > head(data) counts outcome treatment 1 18 1 1 2 17 2 1 3 15 3 1 4 20 1 2 5 10 2 2 6 20 3 2 |
また、パッケージggplot2を用いてcountsとoutcomeの経験分布及び箱ひげ図を描画すると 、outcomeの水準によってcountsが変化することが分かります。
1 2 3 4 5 6 7 8 9 10 | library(ggplot2) library(gridExtra) densityPlot <- ggplot(data, aes(x = counts, y = ..density.., colour = outcome, fill = outcome)) densityPlot <- densityPlot + geom_density(stat = "density", position = "identity", alpha = 0.75) densityPlot <- densityPlot + xlim(min(data$counts), max(data$counts)) boxPlot <- ggplot(data, aes(x = counts, y = outcome, fill = outcome)) boxPlot <- boxPlot + geom_boxplot(alpha = 0.75, colour = "gray") grid.arrange(densityPlot, boxPlot) |
上を実行することで、次のような経験分布と箱ひげ図がプロットされます。outcomeが1のcountsは2, 3のものより比較的大きいことが読み取れます。

また、treatmentとcountsの経験分布と箱ひげ図も以下のように引数を変更することでプロットすることができます。
1 2 3 4 5 6 7 8 | densityPlot <- ggplot(data, aes(x = counts, y = ..density.., colour = treatment, fill = treatment)) densityPlot <- densityPlot + geom_density(stat = "density", position = "identity", alpha = 0.75) densityPlot <- densityPlot + xlim(min(data$counts), max(data$counts)) boxPlot <- ggplot(data, aes(x = counts, y = treatment, fill = treatment)) boxPlot <- boxPlot + geom_boxplot(alpha = 0.75, colour = "gray") grid.arrange(densityPlot, boxPlot) |

treatmentが1のcountsは3のものと比べて、ばらつきが非常に小さいことが分かります。
回帰分析の実行
回帰分析を実行する前に、まずdataをトレインデータとテストデータに分けます。後にテストデータを用いた予測をするために、dataを学習用と検証用に分割します。
今回は簡単に前から9個のデータをトレインデータ、残りのデータをテストデータに分割します。
1 2 3 4 5 6 | #トレインデータとテストデータの作成 trainIndex <- seq_len(9) validationIndex <- setdiff(seq_len(nrow(data)), trainIndex) trainData <- data[trainIndex, ] #トレインデータ validationData <- data[validationIndex, ] #テストデータ |
改めて、次の9行から成るデータに対し回帰分析を実行していきます。
1 2 3 4 5 6 7 8 9 10 11 | > trainData counts outcome treatment 1 18 1 1 2 17 2 1 3 15 3 1 4 20 1 2 5 10 2 2 6 20 3 2 7 25 1 3 8 13 2 3 9 12 3 3 |
ポアソン回帰を実行するには、次のように関数glmの引数を与えます。
1つ目の引数formulaと2つ目の引数dataに関しては、関数lmと全く同じです。formulaにはモデルを表す式を代入し、dataには回帰に用いるデータセットを代入します。
これらの引数に加えて、3つ目の引数familyにpoissonを代入することでポアソン回帰ができます。
1 | regression <- glm(counts ~., data = data, family = poisson) |
regressionを参照するとモデルの回帰係数やAICがあるのが分かります。
1 2 3 4 5 6 7 8 9 10 11 | > regression Call: glm(formula = counts ~ ., family = poisson, data = data) Coefficients: (Intercept) outcome2 outcome3 treatment2 treatment3 3.01539 -0.38137 -0.29585 0.01005 0.01005 Degrees of Freedom: 11 Total (i.e. Null); 7 Residual Null Deviance: 11.37 Residual Deviance: 5.782 AIC: 71.31 |
次にこれらの結果をまとめる方法について解説していきます。
次のように、関数summayによって、これらの解析結果をリストの要素としてまとめることができます。
1 | summaryReg <- summary(regression) |
ポイント
summaryRegを参照すると、Coefficientsの欄の(intercept)とoutcome2の一番右に「*」がついているのが分かります。p値が0.05未満であり、統計的に有意である変数であることが言えます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 | > summaryReg Call: glm(formula = counts ~ ., family = poisson, data = data) Deviance Residuals: Min 1Q Median 3Q Max -1.1450 -0.3701 -0.2113 0.6050 1.1397 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) 3.01539 0.13142 22.945 <2e-16 *** outcome2 -0.38137 0.17336 -2.200 0.0278 * outcome3 -0.29585 0.16908 -1.750 0.0802 . treatment2 0.01005 0.17350 0.058 0.9538 treatment3 0.01005 0.17350 0.058 0.9538 --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for poisson family taken to be 1) Null deviance: 11.367 on 11 degrees of freedom Residual deviance: 5.782 on 7 degrees of freedom AIC: 71.315 Number of Fisher Scoring iterations: 4 |
また、解析結果を得る方法についてみていきます。summaryReg$. . . でsummaryReg中の要素を参照することができます。
1 2 3 4 5 6 7 8 9 10 11 12 13 | > coeffs <- summaryReg$coefficients > aic <- summaryReg$aic > coeffs Estimate Std. Error z value Pr(>|z|) (Intercept) 3.01538709 0.1314192 22.9447986 1.660647e-116 outcome2 -0.38136756 0.1733559 -2.1999112 2.781320e-02 outcome3 -0.29584538 0.1690815 -1.7497203 8.016659e-02 treatment2 0.01005034 0.1734964 0.0579282 9.538058e-01 treatment3 0.01005034 0.1734964 0.0579282 9.538058e-01 > aic [1] 71.31494 |
ポイント
上の例では、summaryReg$coefficientsで回帰係数やp値などが格納されたデータフレームに取得し、summaryReg$aicでモデルのaicを取得しています。
最後に、これらの解析結果をcsvファイルに出力しましょう。
関数write.csvを用いて、上で取り上げた解析結果をcsvファイルに出力します。
データフレームcoeffsとaicを含むデータフレームresultsを以下のように与えます。
1 2 | results <- as.data.frame(cbind(round(coeffs, 5), aic = c(round(aic, 5), rep("", nrow(coeffs) - 1)))) |
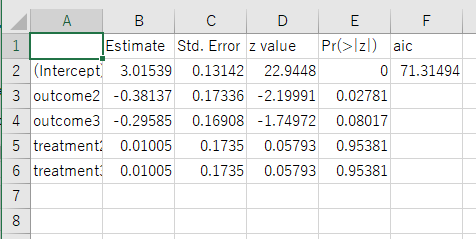
resultsは以下のようになっています。
1 2 3 4 5 6 7 | > results Estimate Std. Error z value Pr(>|z|) aic (Intercept) 3.01539 0.13142 22.9448 0 71.31494 outcome2 -0.38137 0.17336 -2.19991 0.02781 outcome3 -0.29585 0.16908 -1.74972 0.08017 treatment2 0.01005 0.1735 0.05793 0.95381 treatment3 0.01005 0.1735 0.05793 0.95381 |
以下のように、write,csvでデータフレームresultsをcsvファイルへと出力します。
1 | write.csv(results, "ポアソン回帰.csv") |
上を実行すると、作業ディレクトリ中に"ポアソン回帰.csv"が保存されているのが確認できます。
csvファイルを開くと次の画像のように適切に出力されているのが分かります。
テストデータを用いた予測
次に、先ほど作ったテストデータをモデルにあてはめて、テストデータを予測していきます。
テストデータvalidationDataの値をモデルにあてはめるには、関数predictを用います。
次のようにpredictの引数typeを"response"にすることで、予測値を得ることができます。
1 2 3 4 | > predictedY <- predict(regression, validationData, type = "response") #予測値 > predictedY 10 11 12 20.39698 13.92965 15.17337 |
また、予測値とテストデータの比較方法についてみていきます。
これらの予測値を実際の値と比較するために、次のようなデータフレームpredictedDataを構成します。
1 2 3 4 5 6 7 8 9 | > names(predictedY) <- rep("", length(predictedY)) > predictedData <- data.frame(counts = predictedY, + outcome = outcome[validationIndex], + treatment = treatment[validationIndex]) > head(predictedData) counts outcome treatment 1 20.39698 1 1 2 13.92965 2 1 3 15.17337 3 1 |
counts以外のoutcomeとtreatmentはvalidationDataのものと全く同じです。
次に、このpredictedDataとvalidationDataを縦方向に結合させます。
まず、どこまでのデータが予測値であるか実際の値であるかを区別するために、labelsというベクトルを作ります。
1 2 3 4 5 6 | > labels <- c(rep("validation", nrow(validationData)), + rep("prediction", nrow(predictedData))) > labels <- as.factor(labels) > labels [1] validation validation validation prediction prediction prediction Levels: prediction validation |
最後に、rbindとdata.frameを用いて次のようなテストデータと予測値が結合したデータフレームを作成します。
1 2 3 4 5 6 7 8 9 | > validPredData <- data.frame(labels, rbind(validationData, predictedData)) > validPredData labels counts outcome treatment 10 validation 19.00000 1 1 11 validation 16.00000 2 1 12 validation 14.00000 3 1 1 prediction 20.39698 1 1 2 prediction 13.92965 2 1 3 prediction 15.17337 3 1 |
ggplotを用いて、labelsの値についてcountsの値を描画します。
1 2 3 | densityPlot <- ggplot(validPredData, aes(x = outcome, y = counts, fill = labels, color = labels)) densityPlot <- densityPlot + geom_point() densityPlot |
上を実行すると次のようなプロットが出力されます。

青がテストデータの値で、赤が予測値です。
だいたい予測できていることが見て取れます。
誤差構造がベルヌーイ分布(ロジスティック回帰)
次に、誤差構造がベルヌーイ分布に従う場合について考えてみます。
ここでは、二値分類で有名なロジスティック回帰の実行方法を解説していきます。
分類問題で多項ロジットなどもありますが、ここでは目的変数が二値であるものについて説明します。
ロジスティック回帰
ロジスティック回帰は次の線形モデルで表されます。
ここに
また、\(p_i ,\ i = 1, \ldots, n\)に関して次のようにも表される。
0と1の二値から成る目的変数について、\(Y_i=1\)となる確率を\(p_i\)とし、この\(p_i\)に対してロジット変換を行うことで\(-\infty\)から\(\infty\)をとる変数に変換しているのが見て取れます。
難しい話はここまでにして、Rでロジスティック回帰を実行する例をみていきましょう。
データセットの用意
まず、ロジスティック回帰を行うために、パッケージISLRをインストールします。
1 2 | install.packages("ISLR") library(ISLR) |
ISRLには、次のようなDefaultというデータセットが含まれています。
1 2 3 4 5 6 7 8 9 | > data <- Default > head(data) default student balance income 1 No No 729.5265 44361.625 2 No Yes 817.1804 12106.135 3 No No 1073.5492 31767.139 4 No No 529.2506 35704.494 5 No No 785.6559 38463.496 6 No Yes 919.5885 7491.559 |
このデータには"Yes"と"No"から成るdefaultという列があるのが分かります
このdefaultを、ロジスティック回帰を用いて予測していきます。
次に準備として、目的変数defaultの"Yes"と"No"をそれぞれ1と0に変換します。
注意ポイント
1と0に変換した後はfactor型ではなくnumeric型にしましょう。
1 2 3 4 5 | #目的変数を1, 0に変換 data$default <- as.character(data$default) data$default <- replace(data$default, data$default == "Yes", 1) data$default <- replace(data$default, data$default == "No", 0) data$default <- as.numeric(data$default)<> |
ここで、balanceとdefaultをプロットしてみます。次のようにdefaultの値に関してbalanceをプロットしましょう。
1 | plot(data$balance, data$default) |
defaultが0のときbalanceは小さく、defaultが1のときbalanceは大きい傾向があります。

defaultはbalanceによって変化すると考えられるので、defaultをbalanceで回帰することにします。
回帰分析の実行
早速、defaultをbalanceで回帰していきます。
準備として、dataをトレインデータとテストデータに分割します。後にテストデータのあてはめも行いたいので、次のようにtrainDataとvalidationDataという新しい変数を与えます。
1 2 3 4 | trainLabel <- seq_len((3 / 4) * nrow(data)) validationLabel <- setdiff(seq_len(nrow(data)), trainLabel) trainData <- data[trainLabel, ] validationData <- data[validationLabel, ] |
ロジスティック回帰を実行するには関数glmの引数familyを次のように与えます。
1 | regression <- glm(default ~ balance, data = trainData, family = binomial(logit)) |
誤差構造が二項分布であり、リンク関数はロジットであるため、family = binomial(logit)と与えればよいです。
ロジスティック回帰を実行し、regressionを参照すると次のような解析結果が得られます。
1 2 3 4 5 6 7 8 9 10 11 | > regression Call: glm(formula = default ~ balance, family = binomial(logit), data = trainData) Coefficients: (Intercept) balance -10.779111 0.005605 Degrees of Freedom: 7499 Total (i.e. Null); 7498 Residual Null Deviance: 2219 Residual Deviance: 1196 AIC: 1200 |
関数summaryを用いることで、ポアソン回帰と同様に回帰係数やAICを得ることができます。
Coefficientsの欄を見ると、統計検定の結果が載っているのも分かります。(Intercept)とbalanceの一番右に「*」がついているのが分かります。p値が0.05未満であり、これらは統計的に有意な変数であることが言えます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 | > summaryReg Call: glm(formula = default ~ balance, family = binomial(logit), data = trainData) Deviance Residuals: Min 1Q Median 3Q Max -2.3141 -0.1455 -0.0572 -0.0213 3.7053 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -1.078e+01 4.213e-01 -25.58 <2e-16 *** balance 5.605e-03 2.577e-04 21.75 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 2219.0 on 7499 degrees of freedom Residual deviance: 1196.3 on 7498 degrees of freedom AIC: 1200.3 Number of Fisher Scoring iterations: 8 |
summaryRegは回帰係数やAICをリストの要素として持ちます。次のようにして、回帰係数やAICを得ることができます。
1 2 3 4 5 6 7 8 9 10 11 | > summaryReg <- summary(regression) > coeffs <- summaryReg$coefficients > aic <- summaryReg$aic > coeffs Estimate Std. Error z value Pr(>|z|) (Intercept) -10.779110539 0.4213371061 -25.58310 2.352388e-144 balance 0.005604848 0.0002576774 21.75142 6.697852e-105 > aic [1] 1200.347 > |
ポアソン回帰で解説した通り、次のようにして解析結果csvファイルへ出力することができます。
1 2 3 | results <- as.data.frame(cbind(round(coeffs, 5), aic = c(round(aic, 5), rep("", nrow(coeffs) - 1)))) write.csv(results, "ロジスティック回帰.csv") |
モデルを用いた予測
最後に、先ほど作成したvalidationDataをモデルにあてはめていきます。
テストデータの予測値を得るには、ポアソン回帰と同様に関数predictを用います。
1 | predictedY <- predict(regression, validationData, type = "response") |
予測値と実際の値をプロットするために、次のようテストデータと予測値をもつvalidPredDataを作ります。
1 2 3 4 5 6 7 8 9 | > validPredData <- data.frame(validationData, prob = predictedY) > head(validPredData) default student balance income prob 7501 0 No 796.2573 33760.52 1.803520e-03 7502 0 No 506.4623 36596.13 3.559137e-04 7503 0 No 160.0124 48121.18 5.106984e-05 7504 0 No 339.6912 49109.36 1.397951e-04 7505 0 No 0.0000 35288.34 2.082969e-05 7506 0 Yes 1344.7094 17318.84 3.760750e-02 |
validPredDataが作れたら、パッケージtidyverseとggplot2を用いて、ロジスティック回帰曲線とテストデータをプロットします。
1 2 3 4 5 6 7 | library(tidyverse) modelPlot <- ggplot(mutate(predictedData, prob = ifelse(default, 1, 0)), aes(balance, prob)) + geom_point(alpha = 0.15) + geom_smooth(method = "glm", method.args = list(family = "binomial")) modelPlot |
上を実行すると、次の画像が描画されます。

青線がモデルへあてはめたときの値(予測値)で、黒点がテストデータの値です。
balanceが小さいとdefaultは0に近く、大きいと1に近いためある程度予測できていることが分かります。
さらに、判別の精度として誤判別の割合を計算してみましょう。予測値が0.5より大きいか以下であるかでTRUEとFALSEに分割したデータをclassifierとします。
1 2 3 4 | > classifier <- predictedY > 0.5 > head(classifier) 7501 7502 7503 7504 7505 7506 FALSE FALSE FALSE FALSE FALSE FALSE |
次に、関数tableを用いて、defaultの値とclassifierの値に関して2×2の集計表を作成します。
1 2 3 4 5 6 | > classTable <- table(default = validationData$default, classifier = classifier) > classTable classifier default FALSE TRUE No 2409 12 Yes 50 29 |
集計表が作れたら、次のようにして誤判別の割合を計算することができます。
1 2 3 | > missClassProb <- (classTable[2] + classTable[3]) / nrow(validationData) > missClassProb [1] 0.0248 |
0.0248の割合で誤判別をしていることがわかります。このことから、balanceという変数でdefaultをほとんど予測することが可能であることが言えます。
誤差構造がベルヌーイ分布(多重ロジスティック回帰)
最後に、多重ロジスティック回帰について解説します。
さきほどは、defaultという二値変数をbalanceで回帰しましたが、今回はdefaultを他の全ての変数で回帰していきます。
回帰分析の実行
次のように、関数glmのformulaを指定することで簡単に多重ロジスティック回帰を実行できます。
1 | regression <- glm(default ~., data = trainData, family = binomial(logit)) |
関数lmの使いかたで紹介しましたが、formulaを「default ~.」と指定するとdefault以外の変数でdefaultを回帰することができます。
また補足ですが、balanceとincomeでdefaultを回帰したい場合は、次のようにします。
1 | regression <- glm(default ~ balance + income, data = trainData, family = binomial(logit)) |
関数summaryを使って、次のように解析結果をリストに変換することができます。Coefficientsの欄を見ると、(Intercept)とstudentYesとbalanceの一番右に「*」がついているのが分かります。これら3つの変数はp値が0.05未満であり、有意な変数であることがいえます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 | > summaryReg <- summary(regression) > summaryReg Call: glm(formula = default ~ ., family = binomial(logit), data = trainData) Deviance Residuals: Min 1Q Median 3Q Max -2.1972 -0.1396 -0.0538 -0.0190 3.7065 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -1.118e+01 5.811e-01 -19.232 <2e-16 *** studentYes -6.128e-01 2.783e-01 -2.202 0.0277 * balance 5.888e-03 2.739e-04 21.499 <2e-16 *** income 6.131e-06 9.655e-06 0.635 0.5254 --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 2219.0 on 7499 degrees of freedom Residual deviance: 1175.5 on 7496 degrees of freedom AIC: 1183.5 Number of Fisher Scoring iterations: 8 |
解析結果は以下のようにして取り出すことができます。
1 2 3 4 5 6 7 8 9 10 11 12 | > coeffs <- summaryReg$coefficients > aic <- summaryReg$aic > coeffs Estimate Std. Error z value Pr(>|z|) (Intercept) -1.117560e+01 5.811062e-01 -19.2316025 2.013106e-82 studentYes -6.128276e-01 2.782941e-01 -2.2020863 2.765921e-02 balance 5.887594e-03 2.738510e-04 21.4992623 1.581822e-102 income 6.130841e-06 9.654778e-06 0.6350059 5.254246e-01 > aic [1] 1183.546 |
また、これらの解析結果をcsvファイルへ出力したい場合は、次のようにしてできます。
1 2 3 | results <- as.data.frame(cbind(round(coeffs, 5), aic = c(round(aic, 5), rep("", nrow(coeffs) - 1)))) write.csv(results, "多重ロジスティック回帰.csv") |
作業ディレクトリ中に次の"多重ロジスティック回帰.csv"が保存されているのが分かります。
モデルを用いた予測
最後に、モデルの予測性能についてみていきます。
ロジスティック回帰では、balanceでdefaultを単回帰していましたが、今回はdefault以外の変数を全てモデルに組み込みました。
予測性能に変化があったかどうか見ていきます。
単回帰のモデルを用いた予測と同様に、関数predictを用いてvalidationDataの値をモデルにあてはめます。
1 | predictedY <- predict(regression, validationData, type = "response") |
予測値predictedYの値が0.5より大きいか以下であるかでTRUEとFALSEに分類します。これをclassifierとします。
1 2 3 4 | > classifier <- predictedY > 0.5 > head(classifier) 7501 7502 7503 7504 7505 7506 FALSE FALSE FALSE FALSE FALSE FALSE |
次に、関数tableを用いて、defaultの値とclassifireの値に関して集計表を作ります。
1 2 3 4 5 6 | > classTable <- table(default = validationData$default, classifier = classifier) > classTable classifier default FALSE TRUE No 2410 11 Yes 50 29 |
上の集計表から、誤判別の割合を算出してみます。誤判別の割合は次のようにして簡単に求めることができます。
1 2 3 | > missClassProb <- (classTable[2] + classTable[3]) / nrow(validationData) > missClassProb [1] 0.0244 |
0.0244であることから、非常に判別の精度が良いことがいえます。また、単回帰の時と比較すると誤判別の割合が若干減少したことがわかります。
ポイント
このように、モデルに新たな変数を組み込むことで予測精度を向上させることができます。また、AICについても多重ロジスティック回帰の結果の方が小さいことが分かります。
まとめ
R言語で一般化線形モデルを用いた回帰分析を行う方法を解説しました。
目的変数が正規分布に従わない場合に、回帰分析を実行する方法を学びました。
また、テストデータのあてはめ等のモデルへのデータのあてはめの方法も見ていきました。
ぜひ、今回の記事を解析に役立ててください。





