こんにちは、usagi-sanです。
今回は、前回の2標本検定の続きの3群以上の比較方法について解説していきます。
前回の2標本検定の記事が見たい方は、次のリンクをクリックしてください。
-

【R言語】関数t.testを用いた2標本t検定・母平均の差の検定
母平均の差についての検定を行う方法を解説していきます。 R言語を用いることで、母平均の差の検定である2標本t検定が簡単に実行できます。 1標本の時と同様に、検定結果などは自分で計算する必要がなく非常に ...
続きを見る
この記事では、3群以上の比較方法として有名な分散分析について解説していきます。
R言語で分散分析を実行するための関数を紹介するだけでなく、分散分析を行う前提条件や分散分析の方法、その検定のプロット方法、また分散分析後の事後テスト(post-hoc-test)の方法についても紹介します。
この記事に従って、コードを実行すれば一連の分散分析の解析を行うことが可能となっています。
前回のt検定と違い、検定の手順が多く非常にややこしいですが、図を交えて分かりやすく解説していきます。
以下から今回扱うプログラミングコードをダウンロードできます。
また、分散分析の検定方法を詳しく見たい方は次の記事を参照。
-

【統計学】分散分析 一元配置分散分析・二元配置分散分析
複数のグループ間の平均値の検定である分散分析についてみていく。 ここでは固定効果モデルに基づく、繰り返しのある一元配置分散分析と、繰り返しのない2元配置分散分析を解説する。 一元・二元配置分散分析の検 ...
続きを見る
分散分析(ANOVA)の手順
分散分析(ANOVA)の手順を解説していきます。
分散分析には、データの正規性や群間の分散の等質性が前提条件にあったり、分散分析後の事後テストがあったりなど、統計学に詳しくない人にとって、検定が正しく行われているのか判断が難しいことが多いです。
分散分析の実行方法では、それぞれの手順の具体例を与えて、プロットを交えながら、それぞれの検定の役割をみていきます。
分散分析の流れを次の図とステップに示します。

- 分散分析は図の一番上の正規性の検定から始まります。正規性がない場合はノンパラメトリック手法で行います(ノンパラメトリック手法はこの記事では触れない)。
- 次に群間(グループ間)の分散等質性の検定を行う。比較したいグループ間の分散が同等であることを検定します。この条件を満たしていれば次の分散分析が適用できます。分散の等質性が満たされない場合は、Welchの分散分析を適用します(分散が異なる場合はここでは触れない)。
- 分散分析(ANOVA)を行い、群間に差があるかを検定します。ここで、有意な因子(グループを構成するもの、例:品種)が存在するならば、次の事後テストを行います。有意な因子がない場合は、ここで検定を終了し、グループ間に差異はないという結論を得ます。
- 最後に、有意であった因子に対して事後テストを行います。事後テストを行うことでどの群間に差異があるのかを詳しく見ていきます。例えば、品種A、B、Cに分散分析を行ったとき、分散分析では「品種のどれかの間には違いがある」ということしか分かりません。事後テストでは品種A、B、Cのうちどのペア(例AとB間、AとC間)に違いがあるのかを検証することができます。
分散分析の流れは以上になります。
次では、上のステップごとに、R言語で分散分析を行っていきます。
Rスクリプト上でどのステップにいるのかわからなくなった場合は、上の図を活用することをお勧めします。
一元配置分散分析(ANOVA)
パッケージのインストール
分散分析(ANOVA)やその検定のプロットを行う前提として各種パッケージをインストールする必要があります。
次を実行しパッケージのインストールを行い、ライブラリへの追加を行います。
1 2 3 4 5 6 7 8 9 10 11 | install.packages("ggpubr") install.packages("rstatix") install.packages("ggplot2") install.packages("gridExtra") library(ggpubr) library(rstatix) library(ggplot2) library(gridExtra) |
ggpubrは分散分析のプロットに用います。
rstatixは分散分析や事後テストの検定を行うため、またその検定結果をプロット上に表示させるために用います。
これらのパッケージを用いて、次のirisのデータセットを例に分散分析を行っていきます。
1 2 3 4 5 6 7 8 | > head(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 4 4.6 3.1 1.5 0.2 setosa 5 5.0 3.6 1.4 0.2 setosa 6 5.4 3.9 1.7 0.4 setosa |
正規性の検定
まず分散分析の前提として、正規性の検定を行います。
今回irisのSepal.WidthのデータをSpeciesという因子に関して分散分析をします。
Sepal.Widthが正規性を持たない(母集団が正規分布でない)場合、分散分析を適用することができません。
そこでここで紹介する正規性の検定が必須となります。
次のコードを実行することで、正規性に関するプロット及び正規性の検定を行うことができます。
1 2 3 4 5 | #正規性の確認 qqnorm(iris$Sepal.Width ,ylab = "wid") qqline(iris$Sepal.Width) shapiro_test <- shapiro.test(t(iris$Sepal.Width)) |
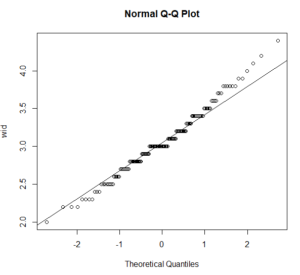
2、3行目を実行すると次のようなプロットが表示されます。
y=xの対角線上にデータのプロットが集中しているほど、データが正規性を持つことがいえます。
上の場合、Sepal.Widthは正規性をもっていることがうかがえます。
プロットだけでは判断できない場合も、5行目を実行することで、正規性の検定であるシャピロ・ウィルク検定を行うことができます。
p値が0.05より大きい場合は正規性をもつことがいえて、次のステップに進むことができます。ここで、p値が0.05以下の場合は、クリスカルウォリス検定などの分散分析のノンパラメトリック手法を検討する必要があります。
実際に次を実行すると、Sepal.Widthに関するシャピロ・ウィルク検定のp値は0.1012>0.05であり、正規性があることが分かりました。
1 2 3 4 5 6 | > shapiro_test Shapiro-Wilk normality test data: t(iris$Sepal.Width) W = 0.98492, p-value = 0.1012 |
正規性の条件が満たされたので、次の群間の分散等質性についてみていきましょう。
群間の分散等質性の検定
正規性の検定に続いて、群間の分散が等しいかどうかを検定していきます。
正規性の検定の時と同様に、分散等質性が満たされない場合、分散分析を適用することができません。
先ほどの正規性の検定の例の続きをみていきます。
群間の分散等質性を検定するには、バートレット検定を行います。
次を実行することで、バートレット検定を行うことができます。
1 2 | #群間の等分散性 bartlett_test <- bartlett.test(iris$Sepal.Width ~ iris$Species) |
barrtlett.test(データ, factor型のベクトル)とすることで、factorのlelves(グループ)ごとのデータの分散が等しいか検定することができます。
bartlett_testの結果を参照すると、次のようにp値0.3515>0.05より、Speciesの各levels(Setosa, versicolor, virginica)のSepal.Widthの分散が等しいことがいえました。
1 2 3 4 5 6 | > bartlett_test Bartlett test of homogeneity of variances data: iris$Sepal.Width by iris$Species Bartlett's K-squared = 2.0911, df = 2, p-value = 0.3515 |
よって、Speciesの群間のSepal.Widthの等分散性がいえたので、分散分析に必要な条件がそろいました。次では分散分析の実行方法、検定結果の見方、結果の保存方法についてみていきます。
分散分析(ANOVA)
分散分析のについて解説していきます。
Sepal.Widthを例に、検定の概要を説明します。
分散分析では、次のような「3群(setosa, versicolor, virginica)間にSepal.Widthの変化はないか」を検定します。
1 2 3 4 | [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11] [,12] [,13] [,14] [,15] [,16] [,17] [,18] [,19] [,20] setosa 3.5 3.0 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 3.7 3.4 3.0 3.0 4.0 4.4 3.9 3.5 3.8 3.8 versicolor 3.2 3.2 3.1 2.3 2.8 2.8 3.3 2.4 2.9 2.7 2.0 3.0 2.2 2.9 2.9 3.1 3.0 2.7 2.2 2.5 virginica 3.3 2.7 3.0 2.9 3.0 3.0 2.5 2.9 2.5 3.6 3.2 2.7 3.0 2.5 2.8 3.2 3.0 3.8 2.6 2.2 |
つまり上の表に関して、Sepal.Widthの標本を\(x_{ij}, i=1,\ldots,50, j=1,2,3\)とすると、これらは次の効果モデルで表現される正規分布の標本であると考えられます。
このとき、次の仮説検定を考えます。
仮説\(H_0\)はSpecies間の効果\(\alpha_j\)に違いがないという仮説を表します。つまり、「Sepal.WidthはSpeciesによって変化しない」という仮説検定を行うことができます。
この仮説検定には次の検定統計量を用います。
ここに、\(S_T\)、\(S_e\)は次で与えられる群間平方和、群内平方和である。
F値が自由度47、2のF分布の有意水準0.05の棄却域に含まれるか否かで、仮設\(H_0\)を棄却または採択します。
では、\eqref{eq1}の仮説検定を実行していきます。
分散分析を実行する関数として、パッケージrstatix中のanova_test(data, formula)を用います。分散分析をするのであれば、他のパッケージを領すれば同様の結果を得ることができます。しかし、他のパッケージではプロットに関する機能が充実していないため、このパッケージを用いた説明をしていきます。
次のように、引数を与えることで\eqref{eq1}の分散分析を実行することができます。
1 2 | #anova anova <- anova_test(data = iris, Sepal.Length ~ Species) |
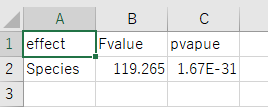
anovaを参照すると次のように、検定結果がリストの中に格納されていることが分かります。p値が1.67e-31<0.05でるため、仮設\(H_0\)は棄却され、Sepal.WidthはSpeciesによって変化するという結果が得られました。
1 2 3 4 5 | > anova ANOVA Table (type II tests) Effect DFn DFd F p p<.05 ges 1 Species 2 147 119.265 1.67e-31 * 0.619 |
次で、anovaの各検定結果を抽出する方法を示しています。
1 2 3 4 5 6 | #検定結果 effect <- anova$Effect DFn <- anova$DFn DFd <- anova$DFd FValue <- anova$F pValue <- anova$p |
- anova$Effectは効果であり、この場合Speciesにあたります。
- anova$DFdは群間平方和\(S_T\)の自由度で、anova$DFdは群内平方和\(S_e\)の自由度です。
- anova$Fは\eqref{eq2}の検定統計量F値です。
- anova$pはこの検定のp値です。
これらの検定結果をcsvファイルへ出力したい値をデータフレームに格納しましょう。
1 2 | #検定結果をデータフレームに格納 one_way_ANOVA <- data.frame(effect = effect, Fvalue = FValue, pvapue = pValue) |
次のようにwrite.csvを実行することで、検定結果をcsvファイルへ出力できます。
1 | write.csv(one_way_anova, "一元配置分散分析.csv") |
出力結果は次のようになります。
以上で一元配置分散分析の解析は終了しました。分散分析の結果、Sepal.WidthはSpeciesによって変化するという結果が得られたため、次ではどのSpeciesのlevels間に違いがみられるかを事後テストを行うことでみていきます。
事後テスト
事後テストでは、分散分析で有意であった効果の各群の比較を行います。
Speciesに関しSepal.Widthの分散分析を行いましたが、分散分析では「 setosa、versicolor、virginicaどこに違いがあるか」ということは分かりません。事後テストではどのsetosa、versicolor、virginicaのペアに違いがあるのかを明確にします。
今回は、事後テストの中でもよく用いられるテューキーの多重比較検定についてみていきます。
テューキーの多重比較検定では次の仮説検定を行います。
分散分析における標本\(x_{ij}\)が\(N(\mu_j , \sigma^2)\)からの観測値であるとき、次の仮説検定を考える。\(k, l = 1,2,3,\ k\neq l\)に対し
テューキーの多重比較検定は次で実行することができます。
1 | pwc <- tukey_hsd(iris, Sepal.Width ~ Species) |
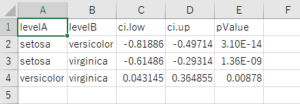
pwcを参照すると、多重比較の検定結果が格納されていることが分かります。group1とgroup2が比較しているペアを表し、1行目はsetosaとversicolorの比較を行っています。p値をみてみるとすべてのp値が0.05未満であるため、すべてのSpecies間に差があることが分かります。またp値の大きさからsetosaとversicolorに一番大きな差異がみられることもわかります。
1 2 3 4 5 6 7 | > pwc # A tibble: 3 x 9 term group1 group2 null.value estimate conf.low conf.high p.adj p.adj.signif * <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> 1 Species setosa versicolor 0 -0.658 -0.819 -0.497 3.10e-14 **** 2 Species setosa virginica 0 -0.454 -0.615 -0.293 1.36e- 9 **** 3 Species versicolor virginica 0 0.204 0.0431 0.365 8.78e- 3 ** |
同様に、csvファイルへ出力するために、これらの検定結果をデータフレームに格納します。
1 2 3 | #データフレームに格納 post_hoc_test <- data.frame(levelA = pwc$group1, levelB = pwc$group2, ci.low = pwc$conf.low, ci.up = pwc$conf.high, pValue = pwc$p.adj) |
次を実行することで、write.csvでcsvファイルへ出力します。
1 | write.csv(post_hoc_test, "Tukey多重比較.csv", row.names = F) |
"Tukey多重比較.csv"を確認すると、次のように検定結果が出力されたことが分かります。
検定結果のプロット
今回の検定の結果をプロットしてみましょう。
多重比較の検定結果を箱ひげ図にプロットし、検定結果を可視化させます。
パッケージggpurとrstatixを用いることで、プロット及びプロット上に検定結果を描画させることができます。
次を実行することで、箱ひげ図に多重比較のp値を記載することができます。
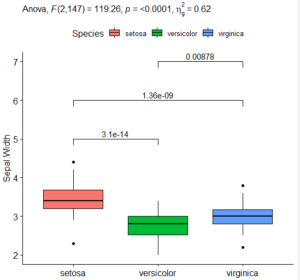
1 2 3 4 5 6 7 | #検定のプロット boxPlot <- ggboxplot(iris, x = "Species", y = "Sepal.Width", fill = "Species") + stat_pvalue_manual(pwc[pwc$term == "Species", ], label = "p.adj", y.position = c(5, 6, 7)) + labs(subtitle = get_test_label(anova[anova$Effect == "Species", ], detailed = TRUE), caption = get_pwc_label(pwc[pwc$term == "Species", ])) boxPlot |
演算子+を用いることで、 1行名のggboxplotの箱ひげ図に、2行目でpwc(多重比較)のp値を加え、3行目以降でプロットのサブタイトルとキャプションを加えています。
箱ひげ図にp値を記載することで、多重比較の検定結果がより把握しやすくなります。
2元配置分散分析
次に2元配置分散分析の実行方法を解説していきます。
1元配置では、「1つのfactor型の因子のデータに関してある変数が変化するか」をみていきましたが、2元配置分散分析では、「2つのfactor型のデータに関して変化するか」をみることができます。
つまり、1元配置には1つの因子データを統計モデルに含めましたが、2元配置では2つの因子をモデルに含めることができより複雑なモデルを基に分散分析を行うことができます。
2元配置分散分析では次のデータセットToothGrouwthを用います。
1 2 3 4 5 6 7 8 9 10 | > #two way anova > data <- ToothGrowth > head(data) len supp dose 1 4.2 VC 0.5 2 11.5 VC 0.5 3 7.3 VC 0.5 4 5.8 VC 0.5 5 6.4 VC 0.5 6 10.0 VC 0.5 |
numeric型であるlenとfactor型であるsuppとdoseを変数を持つことが分かります。
次のようにdata$doseを参照するとfactor型ではないことがわかります。
1 2 3 4 | data$dose [1] 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 1.0 1.0 1.0 1.0 1.0 1.0 1.0 1.0 1.0 1.0 2.0 2.0 [23] 2.0 2.0 2.0 2.0 2.0 2.0 2.0 2.0 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 0.5 1.0 1.0 1.0 1.0 [45] 1.0 1.0 1.0 1.0 1.0 1.0 2.0 2.0 2.0 2.0 2.0 2.0 2.0 2.0 2.0 2.0 |
次のようにして、factor型に変換しましょう。
1 | data$dose <- as.factor(data$dose) |
これでデータセットの準備は整いました。
1元配置の時と同様に、次は正規性や群間の分散等質性についてみていきます。
正規性の検定
次を実行することで変数lenのqqプロットを行うことができます。
1 2 3 | #正規性の確認 qqnorm(data$len ,ylab = "len") qqline(data$len) |
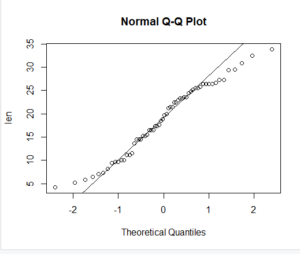
対角線上にデータが分布しているため、正規性があることがうかがえます。
また次を実行することで正規背の検定を行うことができます。
1 2 3 4 5 6 7 | > shapiro_test <- shapiro.test(t(data$len)) > shapiro_test Shapiro-Wilk normality test data: t(data$len) W = 0.96743, p-value = 0.1091 |
p値=0.1091>0.05であるため、正規性があることが言えました。
次は、群間の分散等質性についてみていきます。
群間の分散等質性
2元配置の場合は、各2つのfactor型のデータに対して、群間の分散等質性の検定を行う必要があります。
次のようにして、等質性の検定を実行することができます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 | > #群間の等分散性 > bartlett_test_dose <- bartlett.test(data$len ~ data$dose) > bartlett_test_supp <- bartlett.test(data$len ~ data$supp) > bartlett_test_dose Bartlett test of homogeneity of variances data: data$len by data$dose Bartlett's K-squared = 0.66547, df = 2, p-value = 0.717 > bartlett_test_supp Bartlett test of homogeneity of variances data: data$len by data$supp Bartlett's K-squared = 1.4217, df = 1, p-value = 0.2331 |
変数doseに対して、p値=0.717>0.05であり
変数suppに対して、p値=0.2331>0.05であることから、2つの変数の群間に分散等質性があることが示されました。
分散分析に必要な条件がそろったので、次に2元配置分散分析の実行方法を解説していきます。
分散分析(two-way-anova)
まず、2元配置分散分析の概要を説明します。
次の各factorのlevels別のToothGrowthのデータセットを考えます。
1 2 3 4 5 6 7 | [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] OJかつ0.5 15.2 21.5 17.6 9.7 14.5 10.0 8.2 9.4 16.5 9.7 OJかつ1.0 19.7 23.3 23.6 26.4 20.0 25.2 25.8 21.2 14.5 27.3 OJかつ2.0 25.5 26.4 22.4 24.5 24.8 30.9 26.4 27.3 29.4 23.0 VCかつ0.5 4.2 11.5 7.3 5.8 6.4 10.0 11.2 11.2 5.2 7.0 VCかつ1.0 16.5 16.5 15.2 17.3 22.5 17.3 13.6 14.5 18.8 15.5 VCかつ2.0 23.6 18.5 33.9 25.5 26.4 32.5 26.7 21.5 23.3 29.5 |
つまり上の表に関して、lenの標本を\(x_{ijk}, i=1,\ldots,3,\ j=1,2,\ k=1, \ldots10 \)とすると、これらは次の効果モデルで表現される正規分布の標本であると考えられます。
ただし
このとき、次の2つの仮説検定を考えます。
仮説\(H_{A0}\)はdose間の効果\(\alpha_j\)に違いがないという仮説を表します。つまり、「lenはdoseによって変化しない」という仮説検定を行うことができます。また、仮説\(H_{B0}\)はsupp間の効果\(\alpha_j\)に違いがないという仮説を表します。つまり、「lenはsuppによって変化しない」という仮説検定を行うことができます。
上の2つの2元配置分散分析は、次のように関数anova_testを用いることでできます。引数のformulaにfactor型のデータを"+"で結合させることで、2元配置、3元配置、...を実行することができます。
1 2 | #anova anova <- anova_test(data = data, len ~ dose + supp) |
anovaを参照すると、次のようにdoseとsuppに関する分散分析の結果が格納されていることが分かります。また、p値がそれぞれ6.31e-16<0.05、1.00e-03<0.05であることから、lenはdoseとsuppの両方に関し変化することが分かります。
1 2 3 4 | > anova Effect DFn DFd F p p<.05 ges 1 dose 1 57 123.989 6.31e-16 * 0.685 2 supp 1 57 11.447 1.00e-03 * 0.167 |
これらの検定結果は、次のように1元配置の時と同様に参照することが可能です。
1 2 3 4 5 6 | #検定結果 effect <- anova$Effect DFn <- anova$DFn DFd <- anova$DFd FValue <- anova$F pValue <- anova$p |
次のようにデータフレームに格納することで、csvファイルへの出力が可能です。
1 2 3 4 | two_way_ANOVA <- data.frame(effect = effect, Fvalue = FValue, pvapue = pValue) rownames(two_way_ANOVA) <- rownames(anova[[1]]) write.csv(two_way_ANOVA, "2元配置分散分析.csv", row.names = T) |
事後テスト
事後テストの多重比較については、1元配置の時と同様に関数tukey_hsd を用いることでテューキーの多重比較検定を実行することが可能です。
次のように引数formulaにfactor型のデータを"+"で結合させることで、2元配置、3元配置、...の場合を実行することができます。
1 2 | #多重比較 pwc <- tukey_hsd(data, len ~ supp + dose) |
pwcを参照すると、各levels間の多重比較検定の結果が格納されています。p値はどれも0.05より小さくすべてのlevels間に差異があることが分かります。
1 2 3 4 5 6 7 8 | > pwc #多重比較の結果 # A tibble: 4 x 9 term group1 group2 null.value estimate conf.low conf.high p.adj p.adj.signif * <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> 1 supp OJ VC 0 -3.7 -5.68 -1.72 4.29e- 4 *** 2 dose 0.5 1 0 9.13 6.22 12.0 1.32e- 9 **** 3 dose 0.5 2 0 15.5 12.6 18.4 7.31e-12 **** 4 dose 1 2 0 6.37 3.45 9.28 6.98e- 6 **** |
次のようにして、上の検定結果のデータフレームへの格納とcsvファイルへの出力が可能です。
1 2 3 4 5 6 | #データフレームに格納 post_hoc_test <- data.frame(factor = pwc$term, levelA = pwc$group1, levelB = pwc$group2, ci.low = pwc$conf.low, ci.up = pwc$conf.high, pValue = pwc$p.adj) #csvファイルへの出力 write.csv(post_hoc_test, "2元配置Tukey多重比較.csv", row.names = F) |
検定結果のプロット
最後に検定結果のプロット方法についてみていきます。
事後テストで行った多重比較の検定結果をプロットで表現し、検定結果を視覚的に把握していきます。
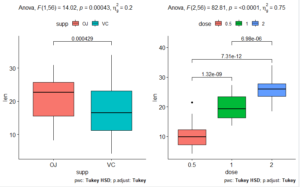
以下を実行することで、変数suppとdoseに関する箱ひげ図とp値の描画ができます。
1 2 3 4 5 6 7 8 9 10 11 | boxPlot_supp <- ggboxplot(data, x = "supp", y = "len", fill = "supp") + stat_pvalue_manual(pwc[pwc$term == "supp", ], label = "p.adj", y.position = 38) + labs(subtitle = get_test_label(anova[anova$Effect == "supp", ], detailed = TRUE), caption = get_pwc_label(pwc[pwc$term == "supp", ])) boxPlot_dose <- ggboxplot(data, x = "dose", y = "len", fill = "dose") + stat_pvalue_manual(pwc[pwc$term == "dose", ], label = "p.adj", y.position = c(30, 36, 42)) + labs(subtitle = get_test_label(anova[anova$Effect == "dose", ], detailed = TRUE), caption = get_pwc_label(pwc[pwc$term == "dose", ])) grid.arrange(boxPlot_supp, boxPlot_dose, nrow = 1) |
上を実行すると次の画像のプロットが描画されます。
2つの変数に関するプロットをgrid.arrangeの引数に代入することで、2つのプロットを描画しています。
検定結果が非常に見やすくなりました。レポートに扱う際に非常に便利です、
まとめ
今回は前回の2群の比較に続いて、3群以上の比較方法についてみていきました。
一元配置分散分析やそれに関係する検定をR言語で実行してみました。
プロット方法を紹介しているサイトが少なかったため、検定のプロットも紹介しました。
分散分析を実行してみたいという方は、是非このソースコードを使ってください。