こんにちは、usagi-sanです。
今回は、データの平均値、分散、相関係数を求めたり、グラフをプロットしたりすることで、データの扱い方や解釈の方法を学んでいきます。
特に相関係数を求めて、グラフを描いてみることは統計解析の初期段階であるため、データを可視化することは非常に重要になってきます。
次のRファイルに今回説明するすべてのプログラミングコードがあるため、コードをコピペして使いたいという場合はダウンロードを推奨します。
データirisを用いた練習
平均値と分散を求める
まず前回も用いたデータirisの平均値と分散を求めてみましょう。
前回の記事にも書いたように、次の関数を用いることでベクトルの平均値と共分散を求めることができます。
1 2 3 | data <- iris mean(data$Sepal.Length) #標本平均値 var(data$Sepal.Length) #不偏標本分散 |
上の例で、dataのSepal.Lengthの標本を\(x_1,x_2,\ldots,x_{150} \)とすると、標本平均はと不偏標本分散は次を計算しています。\begin{align}標本平均\ \ \bar{x}&=\cfrac{1}{150}\sum_{i=1}^{150}x_i\\不偏標本分散\ \ s^2&=\cfrac{1}{149}\sum_{i=1}^{150}(x_i-\bar{x})^2\end{align}
注意として、不偏標本分散は(標本数-1)で割ったものとなっています。
高校で習った分散とは定義が異なるので注意してください。
相関係数を求める
相関係数は関数corを用いることで計算できます。
次のように、異なる2つの変数(データフレームの列)に対して、計算することができます。
1 2 3 | #データの相関係数 cor(data$Sepal.Length, data$Sepal.Width) cor(data$Sepal.Length, data$Petal.Length) |
2行目でdataのSepal.LengthとSepal.Widthの相関係数を求めています。
また、3行目でSepal.LengthとPetal.lengthの相関係数を求めています。
数式で表現すると、Sepal.LengthよSepal.Widthの相関係数とは次を求めています。\begin{align}相関係数: \ \ r = \cfrac{s_{Sepal.L.\ Sepal.W}}{\sqrt{s_{Sepal.L}^2}\sqrt{s_{Sepal.W}^2}}.\end{align}相関係数の性質より、この値の絶対値が大きいほど、2つのデータ間に関連性があることがわかります。
データ解析に用いる主な関数
標本平均値と不偏標本分散などの統計量は以下の関数から計算することが可能です。
| mean(x) | xの平均を求める |
| var(x) | xの分散を求める |
| cov(x,y) | xとyの共分散を求める |
| sd(x) | xの標準偏差を求める |
| cor(x,y) | xの相関係数を求める |
| ave(x) | xの平均を求める |
| max(x) | xの最大値を求める |
| min(x) | xの最小値を求める |
| median(x) | xの中央値を求める |
| quantile(x) | xの最小値, 第1四分位数, 中央値, 第3四分位数, 最大値を求める |
| summary(x) | xの要約統計量(最小値, 第1四分位数, 中央値, 平均, 第3四分位数, 最大値)を求める |
データの標準化
続いて、先ほど求めた標本平均と標本不偏分散を用いて、"Species"以外のデータを標準化してみましょう。
標準化とは次のように、各データから標本平均を引き、標本分散の平方根で割ったものをいいます。(R言語では不偏標本分散の平方根で割っています。)\begin{align}標準化: \ \ \cfrac{x_i-\bar{x}}{s},\end{align}統計学では、便利な性質をもつため、しばしばこの標準化が現れます。
データの標準化には、関数scaleが便利です。
実際に次のようにして"Species"以外のデータを標準化できます。
1 2 3 | #データの標準化 data2 <- data[,setdiff(colnames(data), "Species")]#Species以外のデータ std_data <- scale(data2) |
またfunctionを用いて次のように、標準化の関数を作ることもできます。
1 2 3 4 5 6 7 8 9 10 11 | standarize <- function(data){#実際に作ってみる m <- apply(data, 2, mean) v <- apply(data, 2, var) std <- apply(data, 1, function(data){ (data-m)/sqrt(v) } ) return(t(std)) } std_data2 <- standarize(data2) std_data |
上のコードを上から実行していき、11,12行目の2つの標準化データを見てみると、同じ値を返すことが分かると思います。
データフレームの保存と読み込み
txtファイルへの保存
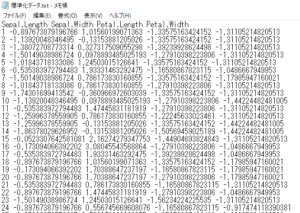
作成したデータフレームをtxtファイルへ保存したい、または読み込みたい場合は以下のように、関数write.tableを用います。
1 2 3 | #データフレームをtxtファイルに保存 write.table(std_data, "標準化データ.txt", col.names = T, quote = F, append = F) #標準化データ.txtとして保存 read.table("標準化データ.txt") #再読み込みできる |
関数write.tableの引数を次の表にまとめました。より詳細なデータの保存が可能となります。
| row.names | TRUEの場合、行名を表示させる |
| col.names | TRUEの場合、列名を表示させる |
| sep | データの区切り文字を指定する。タブ区切りは"\t"、カンマ区切りは","とする。 |
| quote | TRUEの場合、データをダブルクォーテーション""で囲む。 |
| append | TRUEの場合、行末に出力される。FALSEの場合は上書き保存される。 |
| fileEncoding | エンコーディングの指定。WindowsとMacで作業する場合、fileEncoding="CP932"とするとよい。 |
| na | 欠損データNAを指定して文字で置換する。 |
| dec | 小数点を指定した文字で置換する。 |
出力結果は以下の画像のようになります。
csvファイルへの保存
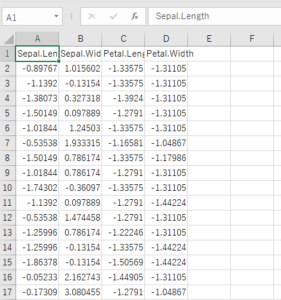
作成したデータフレームは次のように、csvファイルに保存することが可能です。
1 2 3 | #データフレームをcsvファイルに保存 write.csv(std_data, "標準化データ.csv", quote=F, row.names=F, fileEncoding="CP932") #csvファイルへ保存 read.csv("標準化データ.csv", fileEncoding="CP932") #csvファイルを読み込む |
csvファイルに保存したい場合は、write.tableではなくwrite.csvが便利です。
下の画像のように、"標準化データ.csv"をエクセルで確認することができます。
関数の引数は上で紹介したwrite.tableと同じなので省略します。
write.tableと同様に、csvファイルへの詳細な出力の設定が可能となります。
エクセルファイルへに保存
また、エクセルファイルに保存したい場合は、パッケージopenxlxsxをインストールする必要があります。
次のようにすることで結果をexcelファイルに保存することができます。
1 2 3 4 5 6 7 | install.packages("openxlsx") library(openxlsx) wb <- createWorkbook() addWorksheet(wb, "Sheet 1") writeData(wb, sheet = 'Sheet 1', x = std_data, colNames = T , withFilter=F) modifyBaseFont(wb, fontName = "游ゴシック", fontSize = 11,fontColour="#000000") saveWorkbook(wb, "標準化データ.xlsx", overwrite = TRUE) |
上のようにしてエクセルファイルへ保存することができます。
write.xlsxでもできますが、フォント"calibri"が標準となってしまいます。
フォントを変更したい場合は、6行目のように、フォントを"游ゴシック"に変更する必要があります。
データのプロット
関数plot
データのグラフを描きたいとき、関数plotを用いることでをデータをプロットすることが可能です。
1 2 3 4 5 6 7 8 | #データのプロット plot(data2$Sepal.Length, ) #Sepal.LengthとPetal.Lengthのプロット plot(data2$Sepal.Length, xlab = "標本番号", ylab = "length", col = "blue", main = "Lengthのプロット", ylim = c(1,8)) par(new=T) plot(data2$Petal.Length, xlab = "標本番号", ylab = "length", col = "red", ylim = c(1,8)) legend("bottomright", legend = c("Sepal.Length", "Petal.Length"), pch = c(1,1), col = c("blue", "red")) |
上の実行すると、下の画像のプロットを描くことができます。
2行目の実行結果は1枚目の画像であり、Sepal.Lengthのみのプロットとなっています。
横軸は標本のインデックス(エクセルファイルでいう行番号)に対応しています。
4行目以降は、引数legendやpar(new=T)を行うことで、複数のプロットを組み合わせています。

plotの画像

複数のplotを重ねる
関数plotの引数の設定を変更することで上の画像のように詳細な設定を行うことが可能です。
下の表に、引数をまとめました。
| main | タイトル名を指定する | xlim | x軸の範囲を設定する |
| sub | サブタイトル名を指定する | ylim | y軸の範囲を設定する |
| xlab | x軸の名前を指定する | xaxs | FLASEの場合、x軸を描かない |
| ylab | y軸の名前を指定する | yaxs | FLASEの場合、y軸を描かない |
| tmag | タイトルなどのテキストの大きさの倍率 | log | log="x"とすることでx軸を対数軸にする。log="y"、log="xy"も同様に対数軸にする。 |
| col | プロットの点の色を指定する(col = "red", col = "blue"など) | cex | テキストの大きさを拡大縮小する。(cex.axis, cex.lab, cex.mailなどのように細かく指定できる。) |
| adj | テキストの位置(adj = 0: 左揃え, 0.5: 中央揃え, 1:右揃え) | ps | テキストをptx単位で大きさを指定できる。 |
legendの使いかたも下の表にまとめました。複数のグラフを比較するときに重宝します。
| legend | データの種類の名前を指定する |
| pch | プロットの点の種類を変更する |
| col | pchで指定した記号の色を指定する(上の画像の赤い丸の形に指定できたりする) |
データの相関図
下のようにデータフレームをplotに適用すると、データ(Sepal.Legth, Sepal.Width, Petal.Length, Petal.Width)の全ての組み合わせの相関図を描くことが可能です。
1 2 | #データの相関図 plot(data2) |
上のコードを実行すると下の画像を出力する子ができます。

全てのデータの相関図を描く
関数boxplot(箱ひげ図)
関数boxplotを用いることで、箱ひげ図を描くことが可能です。
1 2 3 4 | #箱ひげ図 boxplot(data2) plot(data$Species, data$Sepal.Length , xlab = "", ylab = "") #plotを用いた箱ひげ図の描き方 |
下のコードの2行目のように、data2(Species以外のデータ)をに対してboxplotを行うと、下の画像の箱ひげ図が描かれます。

箱ひげ図
Sepal.LengthをSpecies別で箱ひげ図で描きたい場合、4行目のようにすることで下の画像の箱ひげ図を描くことができます。
ある1つのデータを水準ごとに箱ひげ図を描きたい場合に便利です。

箱ひげ図(Speciesごと)
さらに、boxplotの引数を設定することで下の画像のような、お洒落な箱ひげ図を描くことができます。
1 2 | #boxplot装飾 boxplot(data2, names=c("A", "B", "C", "D"), col=c("#993435", "#edae00", "#539952", "#000000"), main="Boxplot", xlab="Value", ylab="Entry", ylim=c(0, 10), width=c(1.1, 1.1, 1.1, 1.1), staplewex=0.8, horizontal=T, border="cyan4", notch=T, range=1) |

boxplot引数変更
上で用いたboxplotの関数の引数を次の表にまとめました。
ほとんどの引数がplotと同じように指定することができます。
より詳細な箱ひげ図を描くことができます。
| main | タイトル名を指定する | xlim | x軸の範囲を設定する |
| sub | サブタイトル名を指定する | ylim | y軸の範囲を設定する |
| xlab | x軸の名前を指定する | horizontal | TRUEの場合、箱ひげ図を横に描く |
| ylab | y軸の名前を指定する | border | 箱の枠線の色と髭の色を指定する |
| col | プロットの点の色を指定する(col = "red", col = "blue"など) | notch | TRUEの場合、箱に切り込みを入れる |
| width | 箱の横幅の長さ | range | ひげの位置を指定する。デフォルトでは1.5となっている(第3四分位数+1.5×(第3四分位数-第1四分位数の1.5をデフォルトとしている。) |
| staplewex | ひげの幅 |
プロットの保存方法
プロットの保存方法を紹介します。
Rstudioを使っている場合、次の画像のplot画面から、赤矢印のexportを行うことで画像の保存をすることが可能です。

画像保存方法
いちいちエクスポートするのが面倒だという場合は、次のpng("画像目.png")を用いることで、プロットの保存ができます。
plotを完了したら、4行目のdev.off()とすることで無事、作業しているディレクトリにpngファイルが保存されます。
1 2 3 4 | #プロットの保存の仕方 png("プロット画像.png",width = 600 , height = 600) plot(data$Species, data$Sepal.Length , xlab = "", ylab = "") #plotを用いた箱ひげ図の描き方 dev.off() |
データの相関係数
さきほどのプロットで、データの相関を見ることができました。
より数値的に各データの相関を計算したい場合には、cov2corを用います。
この関数は共分散行列を相関行列に変換する関数となっています。
共分散行列を詳しく知りたい方は平均ベクトルと共分散行列#1を見てください。
まず、分散を求めた時に用いた関数varを用いることで共分散行列を次のように計算できます。
1 2 3 4 5 6 | > var(data2) Sepal.Length Sepal.Width Petal.Length Petal.Width Sepal.Length 0.6856935 -0.0424340 1.2743154 0.5162707 Sepal.Width -0.0424340 0.1899794 -0.3296564 -0.1216394 Petal.Length 1.2743154 -0.3296564 3.1162779 1.2956094 Petal.Width 0.5162707 -0.1216394 1.2956094 0.5810063 |
さらに次のように、cov2corを用いることで、共分散行列を相関行列に変換することが可能です。
1 2 3 4 5 6 | > cov2cor(var(data2)) Sepal.Length Sepal.Width Petal.Length Petal.Width Sepal.Length 1.0000000 -0.1175698 0.8717538 0.8179411 Sepal.Width -0.1175698 1.0000000 -0.4284401 -0.3661259 Petal.Length 0.8717538 -0.4284401 1.0000000 0.9628654 Petal.Width 0.8179411 -0.3661259 0.9628654 1.0000000 |
出力結果の見方ですが、上の1行目 2列目はSepal.LengthとSepal.Widthの相関係数、1行目 3列目はSepal.LengthとPetal.Lengthの相関係数,...という風になります。
また、次の自作関数を用いてすべてのirisの4つの変数の相関係数を計算することが可能です。
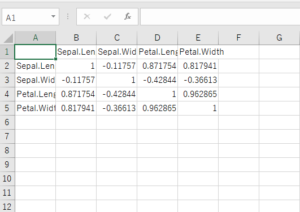
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 | #相関係数 cor(data$Sepal.Length, data$Petal.Length) #Sepal.LengthとPetal.Lengthの相関係数 cor_mat_fun <- function(data){ #相関行列 mat <- matrix(nrow = ncol(data), ncol = ncol(data)) for(i in 1:ncol(data)){ for(j in 1:ncol(data)){ mat[i,j] <- cor(data[,i], data[,j]) } } return(mat) } cor_mat <- cor_mat_fun(data2) cor_mat colnames(cor_mat) <- colnames(data2) cor_data.frame <- data.frame(cor_mat, row.names = colnames(data2)) write.csv(cor_data.frame, "相関行列.csv", append=F , quote=F, sep=",", row.names=T, col.names=T) |
このcor_matは、先ほど求めた相関行列と一致します。
最後の行で、このcor_matをcsvファイルに保存しており、出力結果は次のようになります。