R言語で生存時間解析を行う方法を紹介していきます。
生存率の推定方法の一種であるカプランマイヤー曲線のプロットやCoxの比例ハザードモデル、ログランク検定
を解説していきます。
今回扱うプログラミングコードは以下からダウンロードできます。
生存時間解析
R言語で生存時間解析を行うためにはパッケージsurvivalをインストールする必要があります。
重回帰分析などのように標準で関数がインストールされているわけではないので注意が必要です。
早速、以下のコードを実行しsurvivalをインストールします。
1 2 | install.packages("survival") library(survival) |
パッケージsurvivalにはcancerという癌に関するデータセットが含めれています。時間(time)やイベント発生の有無(status)などの変数があり、まさに生存時間解析の練習用のデータセットといった感じです。
1 2 3 4 5 6 7 8 | > head(cancer) inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss 1 3 306 2 74 1 1 90 100 1175 NA 2 3 455 2 68 1 0 90 90 1225 15 3 3 1010 1 56 1 0 90 90 NA 15 4 5 210 2 57 1 1 90 60 1150 11 5 1 883 2 60 1 0 100 90 NA 0 6 12 1022 1 74 1 1 50 80 513 0 |
このデータセットは既にデータ整形されているため、少し加工し実際のデータに近い形にします。(生存時間や生存と死亡のフラグが既にnumeric型として扱えますが、実際はcharacter型だったり、日付のままだったりするので解説のために加工していきます。)
以下を実行すると、cancerに観測開始日時と終了日時が追加されたデータセットを作成することができます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 | #日付データの生成 tempCancer <- cancer tempCancer$sex <- factor(tempCancer$sex, levels = c(1, 2), labels = c("male", "female")) tempCancer$status <- factor(tempCancer$status, levels = c(1, 2), labels = c("survived", "dead")) dateRange <- list(min = 18000, max = 19000) dateSerial <- sample(dateRange$min : dateRange$max, nrow(tempCancer)) time_start <- as.Date(dateSerial, origin = "1970-01-01") time_end <- as.Date(dateSerial + tempCancer$time, origin = "1970-01-01") tempCancer <- tempCancer[, -seq_len(ncol(tempCancer))[colnames(tempCancer) == "time"]] tempCancer <- cbind(tempCancer, start = time_start) tempCancer <- cbind(tempCancer, end = time_end) tempCancer <- cbind(id = seq_len(nrow(tempCancer)) , tempCancer) dataset <- tempCancer |
以下のように、生存時間ではなく開始と終了に関する日時が追加されているのが分かります。また、生存・死亡のフラグであるstatusも0, 1から"survived", "dead"に変更しました。
1 2 3 4 5 6 7 8 | > head(dataset) id inst status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss start end 1 1 3 dead 74 male 1 90 100 1175 NA 2019-10-09 2020-08-10 2 2 3 dead 68 male 0 90 90 1225 15 2021-10-21 2023-01-19 3 3 3 survive 56 male 0 90 90 NA 15 2021-08-18 2024-05-24 4 4 5 dead 57 male 1 90 60 1150 11 2020-11-03 2021-06-01 5 5 1 dead 60 male 0 100 90 NA 0 2020-04-24 2022-09-24 6 6 12 survive 74 male 1 50 80 513 0 2020-08-29 2023-06-1 |
多分、実務でデータ入力する際に開始日と終了日の差を考慮しないことがほとんどなので、上のようなデータ日付データが一般的だと思います。
生存時間のプロット
まず、生存時間のグラフの描き方についてみていきます。
生存時間解析の本でよく見かける観測開始時点と終了時点に関するグラフを描いていきます。
生存時間を取得する際に用いるDateクラスは次の記事で紹介しています。基本的な日付・時間に関する操作をまとめています。
-

R言語 日付・時間 DateクラスとPOSIXクラス
R言語の日付と時間に関してよく使うテクニックをまとめました。 Rに標準でインストールされているパッケージbaseの中のDateクラスとPOSIXクラスの操作方法について解説していきます。 日付や時間か ...
続きを見る
まず、パッケージggplot2をインストールします。Rのbaseに標準で入っているplotを使うとx軸とy軸を反転できないため、ggplot2を使って描きます。
1 2 | install.packages("ggplot2") library(ggplot2) |
観測開始時点と終了時点に関するグラフは次のようにしてプロットすることができます。変数statusによって色を分けるように指定しているので、死亡("dead")が青で打ち切り("survived")が赤に色分けされています。
1 2 3 4 5 6 | ggplot(tempData) + geom_segment(aes(x = id, xend = id, y = start, yend = end), color = rgb(0, 0, 0, 0.5)) + geom_point(aes(x = id, y = end, color = status), size = 2) + geom_point(aes(x = id, y = start),color = rgb(0, 0, 0, 0.5), size = 2) + coord_flip() + labs(x = "id", y = "time (day)") |

また、グラフのy軸のstartを0に設定すると次のような左端を固定したグラフを描くこともできます。
1 2 3 4 5 6 7 | tempData <- dataset[seq_len(30), ] tempData <- cbind(tempData, time = as.numeric(as.Date(tempData$end) - as.Date(tempData$start))) ggplot(tempData) + geom_segment(aes(x = id, xend = id, y = 0, yend = time), color = rgb(0, 0, 0, 0.5)) + geom_point(aes(x = id, y = time, color = status), size = 2) + coord_flip() + labs(x = "id", y = "time (day)") |

カプランマイヤー曲線
カプランマイヤー法を用いて生存率を推定していきます。
カプランマイヤー法を実行するにはパッケージsurviveの関数survfitを用います。
関数survfit
関数survfitとその引数を以下にまとめます。
| 引数 | 説明 |
| formula | Survオブジェクトまたはcoxphオブジェクト。他のformulaと同様に目的変数を~の左側に、説明変数を右側に指定する。 |
| data | 引数formula、subset、weightsで列名を指定する際にどの列なのかを解釈するためのデータフレーム |
| weights | 非負の重み。正で与えることを推奨している。 |
| subset | モデルを適合する際にデータのどの行を用いるかを指定する。 |
| na.action | 欠損値に対して実行する関数。デフォルトでoptions()$na.actionを行う。 |
| newdata | formulaで指定した変数と同じ変数を持つデータフレーム。formulaがcoxphオブジェクトである場合に適用できる。 |
| individual | logical型。データの行がぞれぞれ異なる個々の時間を表す場合TRUE、個々が複数の時間を持つ場合はFALSE。 |
| conf.int | 信頼区間の信頼水準を指定する。 |
| se.fit | logical型。標準誤差を計算するかどうか。 |
| type | character型。どの手法の生存曲線を求めるかどうかを指定する。"kaplan-meier"でカプランマイヤー法、"fleming-harrington"でfleming-harrington法、"fh2"でfh2法。 |
| error | 誤差の計算方法。"greenwood"でGreenwoodの式、"tsiatis"でTsiatisの式によって計算する。 |
| conf.type | 信頼区間の計算方法。"none"で信頼区間を計算しない。デフォルトで"log"。 |
| conf.lower | 信頼区間の下限の設定。 |
実行例
関数survfitを使ってカプランマイヤー曲線をプロットするコードを紹介します。
まず、さきほど作ったdatasetに生存時間の変数を追加します。観察開始日と終了日の変数は日付を表すcharacter型となっていますが、as.Dateを使ってDate型に変換し、さらにas.numericによってnumeric型に変換することで数値に変換することができます。
1 2 3 4 5 6 7 8 9 | > dataset <- cbind(dataset, time = as.numeric(as.Date(dataset$end) - as.Date(dataset$start))) > head(dataset) id inst status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss start end time 1 1 3 dead 74 male 1 90 100 1175 NA 2019-10-09 2020-08-10 306 2 2 3 dead 68 male 0 90 90 1225 15 2021-10-21 2023-01-19 455 3 3 3 survive 56 male 0 90 90 NA 15 2021-08-18 2024-05-24 1010 4 4 5 dead 57 male 1 90 60 1150 11 2020-11-03 2021-06-01 210 5 5 1 dead 60 male 0 100 90 NA 0 2020-04-24 2022-09-24 883 6 6 12 survive 74 male 1 50 80 513 0 2020-08-29 2023-06-17 1022 |
datasetの一番右の列にtimeが追加されているのが確認できます。
関数survfitを使ってstatus別の生存曲線を描く前に、変数statusとsexをnumeric型に変換する必要があります。
注意ポイント
1 2 3 | survData <- dataset[, c("time", "status", "sex")] survData$status <- as.numeric(as.character(factor(survData$status, levels = c("survived", "dead"), labels = c(0, 1)))) survData$sex <- as.numeric(as.character(factor(survData$sex, levels = c("male", "female"), labels = c(0, 1)))) |
変数statusとsexの整形が完了したら、survfit実行してみましょう。次のように記述することでカプランマイヤー法で生存率を推定することができます。
1 2 3 4 5 6 | > fit <- survfit(Surv(time, status) ~ 1, data = survData, type = "kaplan-meier") #カプランマイヤー曲線 > fit Call: survfit(formula = Surv(time, status) ~ 1, data = survData, type = "kaplan-meier") n events median 0.95LCL 0.95UCL [1,] 228 165 310 285 363 |
他の解析法と同様にfitには生存率やその信頼区間などの変数が含まれており、fitの後ろに$を付けることで参照することができます。
1 2 3 4 | surv <- fit$surv #生存率 lower <- fit$lower #生存率の下限 upper <- fit$upper #生存率の上限 time <- fit$time #生存時間(タイは1つとしている) |
データフレームにこれらの値を代入することで次の画像のように、csvファイルに生存率とその信頼区間を出力することができます。
1 2 | resultTable <- data.frame(surv = surv, ci.lower = lower, ci.upper = upper) write.csv(resultTable, "生存率 カプランマイヤー法.csv") |
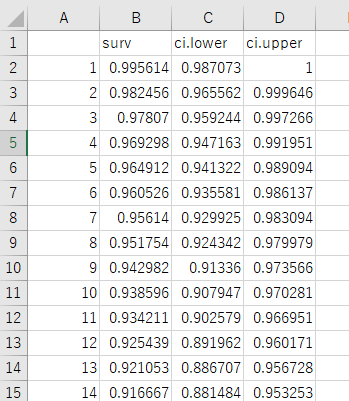
カプランマイヤー生存曲線をプロットする際は、survivalパッケージに含まれている関数plot.survfitを使います。実際に使用する際にはplot()のように使うため、一見、Rのbaseに標準で実装されている関数plotだと思ってしまいますが、こちらの関数は第一引数にSurvオブジェクトを指定するため全く別物となります。
下記のコードを実行することで、通常のカプランマイヤー曲線とsex別のカプランマイヤー曲線が描けます。
1行目のように引数formulaをSurv(time, status) ~ 1と指定することで、グループ別に分けることなく生存曲線を描くことが可能です。factor型の変数のlevelごとに曲線を描きたいときはSurv(time, status) ~ sexのようにします。ここでは性別ごとの生存曲線を描いています。
プロットに打ち切りの線を引きたい場合はmark.T = TRUEとし、信頼区間を描きたい場合はconf.int = TRUEとします。(画像参照)
1 2 3 4 5 6 7 8 9 10 11 | fit <- survfit(Surv(time, status) ~ 1, data = survData, stype = 1, ctype = 1) #カプランマイヤー曲線 fit_sex <- survfit(Surv(time, status) ~ sex, data = survData, stype = 1, ctype = 1) #カプランマイヤー曲線 (sex別) ltypes <- seq_len(nlevels(dataset$sex)) colors <- seq_len(nlevels(dataset$sex)) legends<- levels(dataset$sex) plot(fit, xlab = "time", ylab = "S(t)", mark.t = TRUE, conf.int = TRUE) plot(fit_sex, xlab = "time", ylab = "S(t)", mark.t = TRUE, conf.int = TRUE, col = colors, lty = ltypes) legend(x = "bottomleft", legend = legends, col = colors, lty = ltypes) |


Coxの比例ハザードモデル
続いてCoxの比例ハザードモデルの実行方法について解説します。
Coxの比例ハザードモデルを実行するには関数coxphを用います。
関数coxph
以下、関数coxphとその引数です。
| 引数 | 説明 |
| formula | Survオブジェクト。他のformulaと同様に目的変数を~の左側に、説明変数を右側に指定する。 |
| data | 引数formula、subset、weightsで列名を指定する際にどの列なのかを解釈するためのデータフレーム。 |
| weights | numeric型のベクトル。モデリングの際の重み。 |
| subset | モデルを適合する際にデータのどの行を用いるかを指定する。 |
| na.action | 欠損値に対して実行する関数。デフォルトでoptions()$na.actionを行う。 |
| init | numeric型のベクトル。回帰係数を求める際の反復法の初期値。 |
| control | cox.controlオブジェクト。反復法の際の極限やそのほかのオプションを指定する。 |
| tie | 生存時間にtieが存在するときにどのように部分尤度を計算するかどうか。デフォルトはEfronの近似。 "Breslow"でBreslowの近似。"exact"で正確な部分尤度。 |
| singular.ok | logical型。計画行列が共線性を持つ場合にどうするか。TRUEの場合、行列の特定の行が前の行の線形結合であるとき、自動的に無視する。 |
| robust | logical型。ロバスト分散を計算するかどうか。 |
| id | どの行が同じ対象であるかを指定する。1つの対象が2つ以上のデータを持つ場合指定する。 |
| cluster | 観測値をクラスタリングするためのオプション値。 |
| istate | 状態の初期値。 |
| statedata | 多状態モデルを表現する際の別のデータ。 |
| model | logical型。TRUEの場合、モデルを要素ごとに返す。 |
| x | logical型。TRUEの場合、計画行列Xを要素ごとに返す。 |
| y | logical型。TRUEの場合、応答変数Yを要素ごとに返す。 |
| tt | 時間を変換する関数のリスト。 |
| metod | 引数tieの別の名前。 |
| nocenter | 行列Xの列が指定したベクトルの範囲に存在する場合、センタリングしない。 |
実行例
生存率をセンサーデータstatusとその他の説明変数でモデリングしていきます。
- センサーデータ:status
- 説明変数:sex, age, ph.ecog, meal.cal, wt.loss
次のコードを実行することで、上記の変数のCoxの比例ハザードモデルを行うことができます。
1 2 3 4 5 6 | survNames <- list(time = "time", cens = "status") XNames <- c("sex", "age", "ph.ecog", "meal.cal", "wt.loss") survData <- dataset[, c(survNames$time, survNames$cens, XNames)] survData$status <- as.numeric(as.character(factor(survData$status, levels = c("survive", "dead"), labels = c(0, 1)))) fit <- coxph(Surv(time, status) ~., data = survData, method = "efron") |
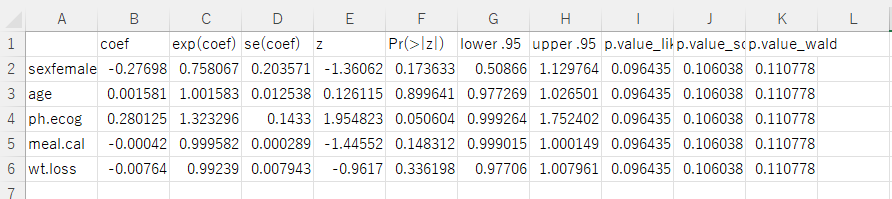
fitを参照すると次のように回帰係数やp値を見ることができます。Likelihood ratio test=9.33 on 5 df, p=0.09643と書いてあることから、timeはsex, age, ph.ecog, meal.cal, wt.lossで変化するとは言えないことが分かりました。また、各説明変数の検定結果からph.ecogのp値が0.0506<0.1であり、timeに影響を及ぼす変数であることがうかがえます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 | > fit Call: coxph(formula = Surv(time, status) ~ ., data = survData, method = "efron") coef exp(coef) se(coef) z p sexfemale -0.2769837 0.7580669 0.2035712 -1.361 0.1736 age 0.0015813 1.0015825 0.0125384 0.126 0.8996 ph.ecog 0.2801255 1.3232958 0.1432996 1.955 0.0506 meal.cal -0.0004181 0.9995819 0.0002893 -1.446 0.1483 wt.loss -0.0076390 0.9923901 0.0079432 -0.962 0.3362 Likelihood ratio test=9.33 on 5 df, p=0.09643 n= 123, number of events= 123 (105 observations deleted due to missingness) |
Coxの比例ハザードモデルの結果を抽出するときは、関数summaryを使うのをお勧めします。
以下のようにsummaryを用いることで、先ほどの回帰係数やp値、各種検定結果等を抽出することが可能です。
1 2 3 4 5 6 7 | summaryFit <- summary(fit) coeffs <- summaryFit$coefficients #回帰係数、標準誤差、p値 confInt <- summaryFit$conf.int #信頼区間 pValue_logtest <- summaryFit$logtest["pvalue"] #尤度比検定 pValue_score <- summaryFit$sctest["pvalue"] #Score検定 pValue_wald <- summaryFit$waldtest["pvalue"] #Wald検定 |
モデルや検定結果などをデータフレームに格納し、csvファイルに出力するには以下のように実行しましょう。
1 2 3 4 5 | resultTable <- cbind(coeffs, confInt[, c("lower .95", "upper .95")], p.value_likelihood.ratio = pValue_logtest, p.value_score = pValue_score, p.value_wald = pValue_wald) write.csv(resultTable, "coxの比例ハザードモデル.csv") |
上を実行すると、次の画像のcsvファイルが作業ディレクトリに出力されます。
ログランク検定
最後にログランク検定についてみていきます。
パッケージsurvivalの関数survdiffを用いることで、ログランク検定を実行することができます。
関数survdiff
以下、関数survdiffとその引数となります。
| 引数 | 説明 |
| formula | Survオブジェクト。Surv(time, status) ~ predictorsのように記述する。 |
| data | 引数formula、subset、weightsで列名を指定する際にどの列なのかを解釈するためのデータフレーム |
| subset | numeric型のベクトル。モデルを適合する際にデータのどの行を用いるかを指定する。マイナスの場合そのインデックスのデータを除外する。 |
| na.action | 欠損値に対して実行する関数。デフォルトでoptions()$na.actionを行う。 |
| rho | numeric型。検定の種類を指定する。 |
| timefix | logical型。関数aeqSurvが丸め誤差を排除する際のい時間を有限にするかどうか。 |
実行例
ログランク検定を用いて性別(sex)によって生存時間(time)が異なるかどうかを検定してみます。
次のように関数survdiffの第一引数にSurv(time, status) ~ sexを代入することで、生存時間が性別によって異なるかというログランク検定を実行することができます。
1 2 3 | survData <- dataset[, c(survNames$time, survNames$cens, "sex")] survData$status <- as.numeric(as.character(factor(survData$status, levels = c("survive", "dead"), labels = c(0, 1)))) testResult <- survdiff(Surv(time, status) ~ sex, data = survData) |
注意ポイント
1 2 3 4 5 6 7 8 9 10 11 | > testResult Call: survdiff(formula = Surv(time, status) ~ sex, data = survData) n=165, 63 observations deleted due to missingness. N Observed Expected (O-E)^2/E (O-E)^2/V sex=male 112 112 102.8 0.824 2.24 sex=female 53 53 62.2 1.362 2.24 Chisq= 2.2 on 1 degrees of freedom, p= 0.1 |
testResultを参照すると、観測度数や期待度数、p値などのカイ2乗検定に関する値がコンソールに表示されます。また、Chisq= 2.2 on 1 degrees of freedom, p= 0.1 より有意水準0.05で生存時間(time)は性別(sex)によって変化するとは言えないことが分かりました。
testResultの後ろに$を付けることで、これらの検定結果の値を取得することができます。
1 2 3 4 5 6 7 8 9 10 | N <- testResult$n #標本数 Obs <- testResult$obs #死亡の観測度数 Exp <- testResult$exp #期待度数 Var <- testResult$var #共分散行列 chisq <- (Obs - Exp)^2 / Exp #カイ2乗統計量 stat_chisq <- testResult$chisq #検定統計量 #p値 pValue <- pchisq(testResult$chisq, nlevels(dataset[, "sex"]) - 1, lower.tail = FALSE) |
Coxの比例ハザードモデルでもやったように、データフレームに代入することで検定結果をcsvファイルに出力することができます。
1 2 3 4 5 | resultTable <- data.frame(N = N, Obs = Obs, Exp = Exp, Chisq = chisq, Stat_chisq = stat_chisq, p.value = pValue) write.csv(resultTable, "ログランク検定.csv", row.names = FALSE) |
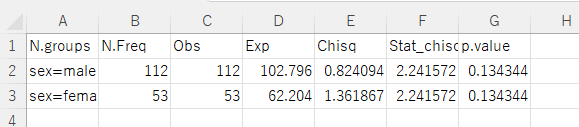
まとめ
R言語で生存時間解析を解説しました。
この記事ではカプランマイヤー曲線や、Coxの比例ハザードモデル、ログランク検定など有名な解析を紹介しました。
今回、関数の引数などの詳細はあまり触れませんでしたが、詳細についてはRdocumentationで調べるのをお勧めします。




